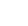近日,深圳北理莫斯科大學計算數學與控制系教授張振躍取得重要科研突破。其作為第一作者、以深圳北理莫斯科大學為第一單位的研究論文《Global understanding via local extraction for data clustering and visualization》,在國際頂級學術期刊《Patterns》(CELL出版社子刊)上發表。該研究聚焦復雜無標簽數據聚類與可視化難題,提出GULE框架,通過類一致的局部提取、全局傳播以及自我學習,實現高精度聚類(如 RNA-seq 數據細胞類型鑒定)和拓撲結構保留可視化,為生物醫學等領域提供新工具,推動多學科數據模式發現。

在當今大數據時代,從復雜數據中提取潛在類別信息是科學研究領域的重要挑戰。無論是生物醫學中的細胞分類,還是社交網絡中的用戶行為分析,傳統的聚類方法往往依賴于對數據結構或分布的強假設,然而,現實數據通常具有高度復雜性,缺乏明確的分布規律,導致現有算法的準確性和魯棒性受限。如何在不依賴預設條件的情況下,從原始數據的局部關聯中挖掘潛在類別,成為亟待解決的問題。
圖1:GULE框架概述
GULE(Global Understanding via Local Extraction)框架基于“局部一致性提取-全局傳播”的核心原理,通過兩層自學習網絡實現類結構解析。該方法通過兩個核心步驟實現:一是局部提取,從數據的局部連接中捕捉類別一致性,無需預先假設數據結構;二是全局傳播,將局部發現的一致性信息通過全局網絡傳遞和自我學習,最終形成完整的類別劃分。研究人員通過理論分析證明,GULE能夠高精度地還原數據中的潛在類別。此外,該方法還可用于數據可視化,在降維過程中保留類別的拓撲結構。實驗表明,GULE在聚類準確性和可視化可靠性上均顯著優于傳統方法,尤其在生物醫學等復雜數據場景中表現突出。

圖2:小鼠腦數據集上的腦細胞聚類方法性能對比
GULE通過三項關鍵技術,為復雜數據處理開辟了新路徑。首先為自適應圖切割(Acut),通過參數β調節類內連接最大化與類間連接最小化的平衡,適應不同密度和結構的數據集。其次是漸進式學習:兩層投影逐步優化類一致性,第一層處理原始數據的稀疏圖,第二層針對低維投影的密集圖進一步細化,提升聚類精度。最后通過拓撲保留可視化,結合t-SNE等技術,將原始數據與GULE投影結合,在降維中保留類內拓撲結構,如COIL20數據集的環狀結構和PIE數據集的線性模式。
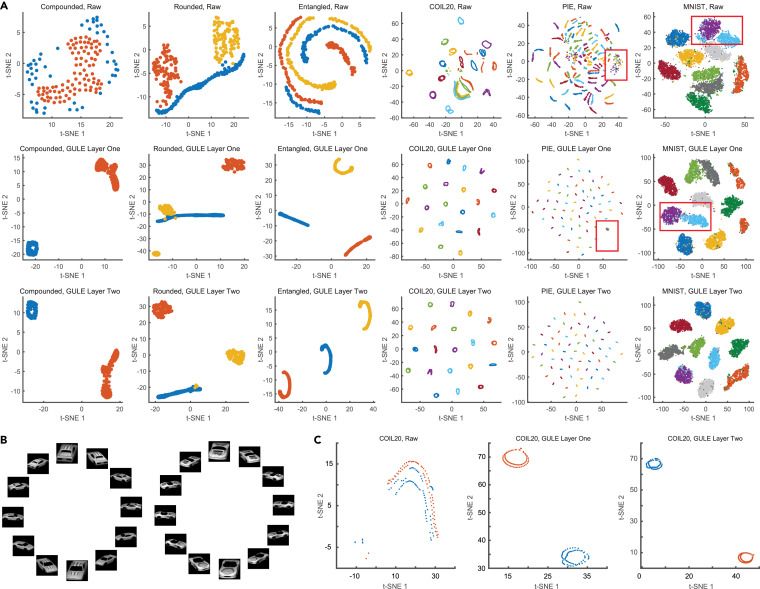
圖3:基于GULE投影的數據可視化拓撲增強
GULE的核心創新在于擺脫了對數據分布的傳統假設,僅借助局部關聯來挖掘全局模式。這一突破性理念為處理現實世界中的非結構化數據開辟了全新路徑。這項研究不僅推動了無監督學習技術的發展,也為跨學科復雜數據分析提供了實用工具。未來,GULE或將成為數據驅動研究的重要基石,為生物學、醫學等領域的多樣化應用提供新見解。
論文鏈接:https://www.cell.com/patterns/fulltext/S2666-3899(25)00114-X